Om voedingsmicrobiologische analyses van kiemgetalbepaling tot detectie sneller, aan lagere kost, gebruiksvriendelijker en met behoud van betrouwbaarheid mogelijk te maken, kunnen we ons meer en meer beroepen op innovaties van laboratoriumschaal tot op moleculair niveau.
Voedingsmicrobiologisch onderzoek vormt de hoeksteen van voedselveiligheids- en controleprogramma’s bij de productie van voedingsmiddelen. Het omvat het bepalen van de aantallen (kiemgetalbepaling) en soms ook van de aanwezigheid en identiteit van micro-organismen die zich ophouden in grondstoffen, processtromen en -producten, eindproducten, verpakte producten en de procesomgeving. Op basis daarvan kan men vaststellen of het proces voedselveilig verloopt of kan men bijvoorbeeld een verificatie doen van het HACPP-systeem. Naast voedingsbedrijven, in het kader van hun autocontrole, voeren ook afnemers, controleorganismen en de overheid microbiologisch onderzoek uit om na te gaan of producten voldoen aan vastgelegde criteria.
Voedingsmicrobiologische analyses worden dus op grote schaal uitgevoerd. Erkende service laboratoria nemen daarvan het grootste aandeel op zich. Omdat klassieke routine-microbiologische analyses tijdrovend (opkweek van micro-organismen op voedingsmedia) en ook nog eens arbeidsintensief zijn, zijn dergelijke laboratoria vragende partij voor alternatieve methoden die sneller, aan lagere kost en gebruiksvriendelijker resultaten kunnen leveren. Voorwaarde is wel dat de methoden op een erkende en gestandaardiseerde manier uitgevoerd worden (bv. volgens een ISO-norm). Dat geldt trouwens ook voor de voorbereidingsstappen.
Innovaties op macroschaal
Enkele voorbeelden van laboratoriuminnovaties die inmiddels ingang gevonden hebben voor het uitvoeren van kwantitatieve plaattellingen (kiemgetalbepalingen in cfu/ml of cfu/g) zijn:
> Het gebruik van chromogene selectieve agarmedia voor specifieke analyse van bijvoorbeeld Bacillus cereus: Deze methode (met AFNOR certificaat AES 10/10-07/10) maakt een twee- tot driemaal snellere analyse mogelijk in vergelijking met de klassieke referentie methode op basis van MYP (‘mannitol-yolk polymyxin’) agar (ISO 7932).
> Het gebruik van spiraalplaatapparaten voor het beënten van petriplaten: Hierbij wordt een vloeibaar monster of verdunning in een spiraalvorm op een agarplaat aangebracht waarbij van binnen naar buiten toe steeds minder vloeistof (en dus ook micro-organismen) wordt aangebracht. Het resultaat is een verdunningsreeks van vier opeenvolgende decimale verdunningen op 1 plaat.
> Het toepassen van automatiseringsoplossingen:
- Staalidentificatie, -registratie en -tracering via barcode toekenning
- Het robotiseren van allerlei handelingen: pipetteren, behandeling van petriplaten,…
- Het automatiseren van petriplaat incubatie en stockage
- Het automatiseren van het tellen van de kolonies op de platen
- Geavanceerde dataverwerking en informatie-extractie
Laboratoria voeren ook kwalitatieve bepalingen uit. Gekende analyses op dat vlak zijn bijvoorbeeld detectietesten om de aan- of afwezigheid van pathogenen zoals bijvoorbeeld Listeria monocytogenes en Salmonella in een bepaalde hoeveelheid (vaak 25 gram) product aan te tonen. Ook hier worden versnelde, gevalideerde methoden uitgevoerd. Een voorbeeld hiervan is de detectiemethode voor Listeria monocytogenes die gebruik maakt van een chromogeen selectief agarmedium in combinatie met een bevestigingstest die uitgevoerd wordt op (enkele) kolonies die op de petriplaat met het selectieve medium tevoorschijn komen:
- Het selectief medium screent op de aanwezigheid van specifieke Listeria monocytogenes fosfolipasen C en op het onvermogen van Listeria monocytogenes om xylose te metaboliseren.
- De bevestigingstest op de kolonies kan snel gebeuren met een moleculaire methode (real-time PCR – zie verder) of met MALDI-TOF.
- De totale analyse neemt 48 tot 56 uur in beslag, tegenover 5 tot 10 dagen met de klassieke detectiemethode (ISO 11290).
Innovaties op nanoschaal
In tegenstelling tot klinische stalen waar het aantal mogelijke matrices beperkt is (veelvoorkomend zijn bijvoorbeeld faeces-, bloed- en urinestalen) en de hoeveelheid pathogenen hoog kan oplopen, is bij voedselstalen de variatie in matrices hoog en de hoeveelheid pathogenen laag. De detectie van voedselgebonden pathogenen in voedselstalen komt dan ook vaak neer op het vinden van een speld in een hooiberg. Het ontwikkelen van moleculaire technieken die in essentie hun werk op nanoschaal uitvoeren en die zich specifiek kunnen richten naar DNA van doelorganismen hebben echter voor een revolutie gezorgd in de voedingsmicrobiologische diagnostiek.
Vooral PCR (polymerasekettingreactie) wordt veelvuldig toegepast. Deze techniek heeft al een lange historiek achter de rug. De Nobelprijs voor Scheikunde werd er in 1993 voor toegekend. Het vereist een DNA extractiestap in de staalvoorbereiding. Om de reactie uit te voeren, is een thermocycler essentieel. Dit is een toestel dat de temperatuurcycli doorloopt die de verschillende rondes van DNA-replicatie regelen. De detectielimiet bedraagt 103-104 cellen per gram of per ml. De techniek bleek al gauw goed toepasbaar op reinculturen en wordt ondertussen standaard ingezet voor bevestigingstesten, bijvoorbeeld voor Listeria monocytogenes (zie eerder).
PCR kan ook toegepast worden op DNA dat geïsoleerd wordt uit voedingsstalen die van nature rijk zijn aan micro-organismen (bijvoorbeeld zuurdesems, ambachtelijke kazen,…). Door universele 16S ribosomale RNA genen te ‘targetten’, bekomt men dan een goed beeld van de samenstelling van het bacteriële ‘microbioom’. Dit is het geheel aan micro-organismen. Deze aanpak is evenwel niet zo geschikt voor het opsporen van pathogene micro-organismen in voeding, gezien de lage hoeveelheden waarbij deze typisch aanwezig zijn (Gérard et al., 2021). Bijgevolg dient er voor het analyseren van voedingsstalen op pathogenen meestal een specifieke aanrijkingsstap (ook ophoping genoemd) te worden toegepast in de staalvoorbereiding. Vandaag de dag staan er commerciële diagnostische kits en systemen voor bijvoorbeeld Salmonella-detectie ter beschikking waarbij het volledige protocol versneld kan uitgevoerd worden. Bovendien zijn een aantal van deze gevalideerd door bijvoorbeeld AFNOR of Microval (GeneDisc® Plate, iQ-CheckTM Salmonella II).
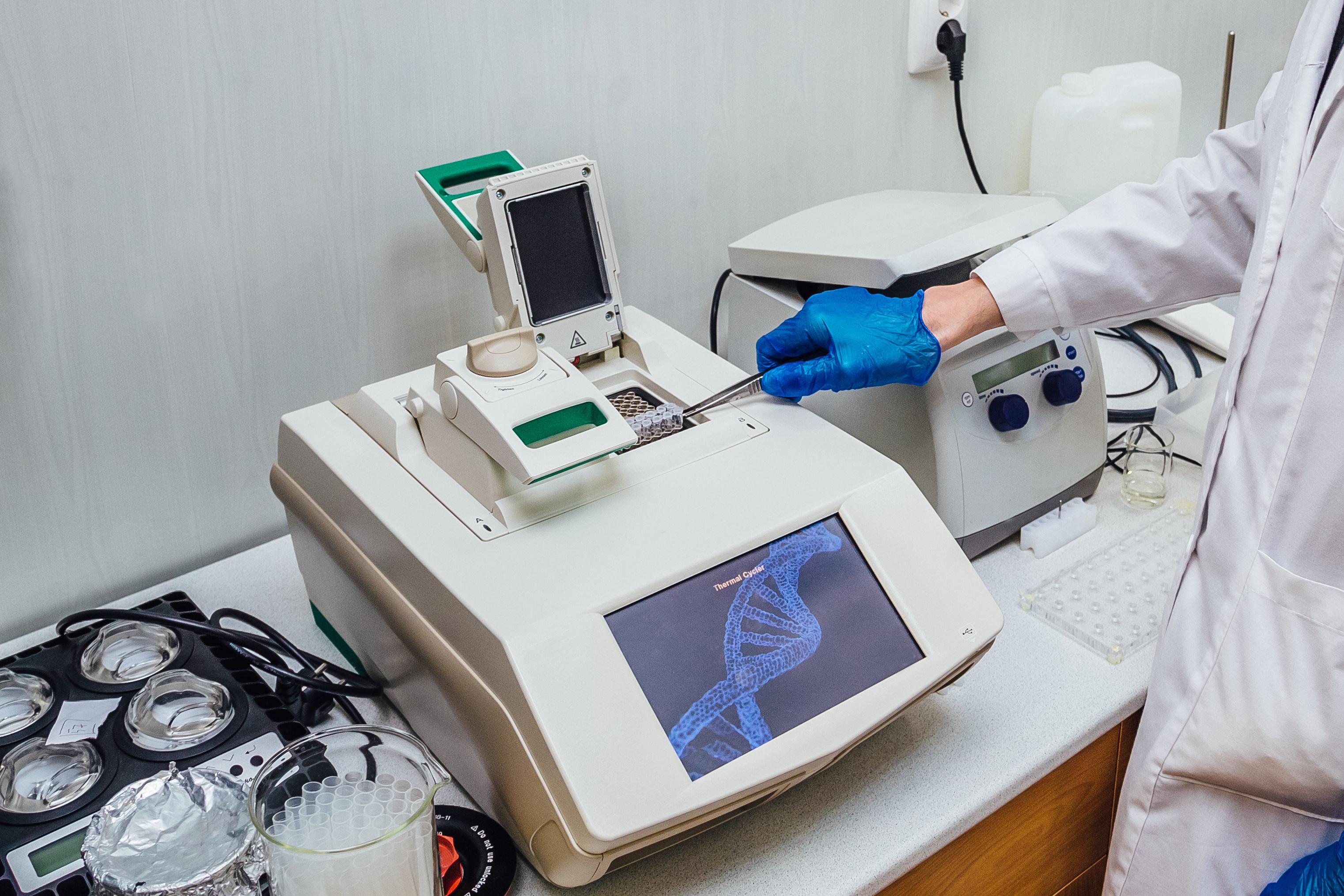
Voor kwantitatieve bepalingen van voedselpathogenen kan kwantitatieve PCR (qPCR) worden toegepast. Hierbij wordt de stapsgewijze vordering van de polymerasekettingreactie (de door temperatuurcycli gedreven DNA-amplificatie) in ‘real-time’ gemonitord via fluorescentie meting. De exponentiële amplificatie (zie onderstaande figuur) gaat sneller op naarmate er meer DNA van het doelorganisme in het staal aanwezig is. Op basis van een standaardcurve kan dan het kiemgetal afgeleid worden.
Een alternatieve kwantitatieve bepaling kan uitgevoerd worden via digitale PCR (dPCR). Hierbij wordt het PCR-staal verdeeld in compartimenten (bv. in minuscule vetdruppeltjes) en wordt in elk daarvan nagegaan of de PCR-reactie opgaat of niet (en dus het ‘target DNA’ aanwezig is of niet). Het aantal targets in het staal kan dan worden afgeleid van de verhouding tussen het aantal positieve druppels ten opzichte van het totale aantal (via binomiale Poisson statistiek).

Modelvoorstelling van een Real-Time qPCR plot. Per cyclus van de PCR-amplificatiereactie wordt de toename in fluorescentie (ΔRn) opgemeten. De PCR-cyclus waarbij de amplificatiereactie zich exponentieel begint op te bouwen, wordt de Ct waarde genoemd (in het voorbeeld is dat cyclus 17). Deze waarde wordt sneller (bij minder aantal PCR cycli) bereikt naarmate er meer DNA van het doelorganisme (o.v.v. een specifieke ‘target’ DNA template) in het staal aanwezig is. [Bron: NCBI]
Een alternatief voor PCR is de LAMP-(Loop mediated isothermal amplification) methode. Hiervoor heeft men geen thermocycler nodig, aangezien de amplificatiereactie (op een eenmalige DNA-denaturatiestap na) bij één welbepaalde temperatuur verloopt (65°C, de optimale temperatuur voor het aangewende Bst DNA polymerase met ‘strand-displacing’ activiteit). De LAMP-reactieproducten zijn lange slierten van aaneengeschakelde kopieën van een specifieke DNA ‘target’ sequentie. Net zoals bij PCR zijn zowel kwalitatieve als kwantitatieve (real-time) analyses mogelijk. LAMP staat bekend als een robuuste methode die zeer snel resultaat geeft (reactie < 1 uur), wat mogelijkheden biedt voor ‘point-of-care-testing’ (POCT, waarbij een diagnostiek onmiddellijk na staalname wordt uitgevoerd) op het terrein (bv. in productie-eenheden van de voedingsindustrie).
Meer en meer komen er geïntegreerde, draagbare systemen op de markt voor POCT-toepassingen en dit zowel PCR-gebaseerd (bv. Bento Lab Portable DNA Laboratory) als LAMP-gebaseerd (bv. 3M™ Molecular Detection System). Daarnaast kunnen ook immunologische methodes ingezet worden voor POCT onder de vorm van Lateral flow immuno-assays. Gedegen validatie van deze testen is echter aangewezen om betrouwbare resultaten te kunnen opleveren. Verdere evoluties in POCT wordt verwacht van onderzoek in ‘microfluidics’ en analyt-detectie (bv. Surface Plasmon Resonance, Surface-Enhanced Raman Scattering, elektrochemische biosensoren,…) die de ontwikkeling van lab-on-chip systemen mogelijk maken. De schaling naar miro en nanoschaal zou snellere reacties en betere sensitiviteit opleveren en dit met minder staal en reagentia. Alles zou ook een stuk geautomatiseerder verlopen. Bottleneck hierbij blijft vooralsnog de staalvoorbereiding (bv. DNA extractie en opzuivering).
In het SIPORE project voeren we daarom onderzoek naar een biosensorsysteem dat rechtstreeks de doelbacterie kan detecteren met een minimum aan staalvoorbereiding (zonder aanrijkingsstap of DNA isolatiestap). Het hart van het systeem bestaat uit elektrochemische SIP biosensoren die een ‘receptorlaag’ (coating) van ‘Surface Imprinted Polymers’ bevatten die gefunctionaliseerd zijn om een specifiek micro-organisme te binden (‘whole-cell receptors’).
Wilt u als bedrijf deelnemen aan SIPORE (als lid van de industriële adviesraad) om het hele onderzoek vanop de eerste rij te volgen? Of wilt uw bedrijf zelf ontwikkelingen starten rond SIP-biosensors? Neem dan zeker contact op met Steven Van Campenhout.
Gebruikte bronnen:
- Event “Nieuwe SIP-biosensor: opportuniteiten voor ‘on site’ microbiologische analyses”:
- Presentatie ‘Evoluties in de klassieke voedingsmicrobiologie toegepast in service labs en voedingsindustrie’ (Koen De Reu, ILVO)
- Presentatie ‘Moleculaire methoden voor snelle detectie in de voedingsmicrobiologie’ (Marc Heyndrickx, ILVO)
- Presentatie ‘Kick-off VLAIO project SIPORE’ (Patrick Wagner, KU Leuven; Marc Heyndrickx, ILVO; Gunther Anthonissen, UGent; Steven Van Campenhout, Flanders’ FOOD)
- Gérard A, El-HajjajiS, Burteau S, Abdoulaye Fall P, Pirard B, Taminiau B, Daubec G and Sindica M. (2021) Study of the microbial diversity of a panel of Belgian artisanal cheeses associated with challenge studies for Listeria monocytogenes. Food Microbiology, 100, 103861.
- Yang L, Yi W, Sun F, Xu M, Zeng Z, Bi X, Dong J, Xie Y and Li M (2021) Application of Lab-on-Chip for detection of microbial nucleic acid in food and environment. Frontiers in Microbiology, 12, 765375.