Met ‘Surface Imprinted Polymers’ (SIP)-biosensoren zijn soortgerichte on site microbiologische analyses mogelijk zonder dat een PCR- of aanrijkingsstap nodig is. Het zopas opgestarte project SIPORE werkt naar voedingsmicrobiologische toepassingen toe en onderzoekt de combinatie met draagbare derde-generatie DNA-sequentieanalyse om zo snel informatie tot op stamniveau te kunnen verkrijgen.
Op 14 juni werd het SIPORE project afgetrapt met een open kickoff-event in de Food Pilot. Het project SIPORE, voluit ‘Surface Imprinted Polymers technology and nanopORE sequencing for point of care food microbial analysis’, zal zich de komende twee jaar focussen op het ontluiken van het potentieel van ‘SIP biosensoren’ en ‘nanopore’ DNA-sequentieanalyse als innovatieve technologiën om voedingsbedrijven in staat te stellen snel, betrouwbaar en robuust soort-specifieke voedingsmicrobiologische analyses in huis uit te voeren. Twee toepassingen worden beoogd: (1) ‘point of care’ screening voor de aan/afwezigheid van pathogene bacteriën en (2) opvolging van specifieke bacteriën (bijvoorbeeld starterculturen) in (complexe) voedingsfermentaties.
De SIP-biosensor vangt specifieke bacteriën op zijn sensoroppervlak via ‘imprints’ die fungeren als een mal. Eerder werd de SIP-biosensor voornamelijk op basis van modelmicro-organismen ontwikkeld en bestudeerd (ondermeer in het agrEUfood project). SIPORE zet de stap naar voedingsmicrobiologische analyses door zich te richten op enkele bacteriën die relevant zijn voor de voedingsindustrie, namelijk: Listeria monocytogenes, Salmonella Enteritidis en S. Typhimurium, Lactobacillus plantarum en, indien de tijd het toelaat, ook Bacillus cereus. Het project werkt niet enkel aan specifieke SIP-biosensoren voor deze organismen maar onderzoekt tegelijkertijd hoe de performantie van de sensoren verbeterd kan worden, zowel ter hoogte van de bioreceptorlaag als via aanpassingen aan het meetprincipe. Een specifiek werkpakket richt zich op het optimaliseren van protocols en een validatie van de specificiteit (voor een bepaalde doelbacterie, op soortniveau), inclusiviteit (alle stammen binnen een doelbacterie), gevoeligheid (het vermogen om doelbacteriën te ‘vangen’ in aanwezigheid van achtergrondmicrobiota), detectielimieten, reproduceerbaarheid en herbruikbaarheid van de biosensorchips.
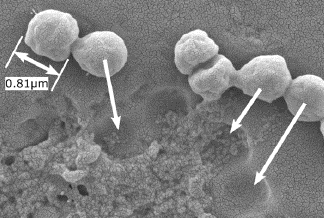
Beeld met Scanning Electron Microscope (SEM) van een SIP-biosensor met imprints voor de coccoïde vorm voor Campylobacter jejuni en de bacteriën zelf (16 000 x vergroting). Bron: Givanoudi S. et al. (2021) Selective Campylobacter detection and quantification in poultry: A sensor tool for detecting the cause of a common zoonosis at its source, Sensors and Actuators B: Chemical, Volume 332, 129484.
Daarnaast is verdere downstream analyse van de op de SIP-sensor gebonden organismen ook mogelijk. In het project zal meer specifiek gekeken worden naar DNA-sequentieanalyse. Eerst zullen de mogelijkheden van tweede- en derde-generatie DNA-sequentieanalyse voor ‘whole genome sequencing’ worden verkend om on site typeringen tot op stamniveau te kunnen uitvoeren. Een bioinformatische workflow zal worden uitgetest om deze typering te kunnen doen, zelfs binnen samengestelde mixen van verschillende stammen. Bijkomend zal de mogelijkheid onderzocht worden om in de gegenereerde DNA-sequentiedata specifieke genoomelementen (bv. extrachromosomale plasmiden) of specifieke genen (bv. biofilmvorming of resistentie tegen antibiotica en desinfectantia) op te sporen. Tot slot wordt de combinatie van de SIP-biosensortechnologie met ‘draagbare’ nanopore sequencing onderzocht met het oog op een geïntegreerd werkschema.
Wilt u als bedrijf deelnemen aan SIPORE als lid van de industriële adviesraad om het hele onderzoek vanop de eerste rij te volgen? Of wilt uw bedrijf zelf ontwikkelingen starten rond SIP-biosensors en/of DNA-sequentieanalyse voor een eigen toepassing of voor een innovatie? Neem dan zeker contact op met Steven Van Campenhout.
Gebruikte bronnen:
- agrEUfood: https://www.agreufood.eu/
- Givanoudi S. et al. (2021) Selective Campylobacter detection and quantification in poultry: A sensor tool for detecting the cause of a common zoonosis at its source, Sensors and Actuators B: Chemical, Volume 332, 129484.
- Event ‘Nieuwe SIP-biosensor: opportuniteiten voor ‘on site’ microbiologische analyses’, 14 juni 2022